앞선 포스팅 Kaplan-Meier Estimation에서 구간생존율을 누적하여 누적생존율을 구하는 방법을 확인했습니다. 의료 분야에서는 생존 분석을 위해 다양한 방법들이 사용됩니다. Kaplan-Meier 방법이 특정 집단의 생존율을 추정하는 방법이라면, 이번 포스팅에서 살펴볼 Log-Rank Test는 두 집단의 생존율의 차이가 유의미하게 있는지 확인하기 위한 방법입니다. 기존의 생존 분석과 마찬가지로 시간(Time)과 사건(Event) 개념이 필요합니다. Log-Rank Test는 두 집단의 데이터를 병합하여 관찰 시간 순으로 정렬하고, 절단 데이터(Censored Data)를 제거합니다. 결과적으로 사건이 발생한 구간만을 남기는 방식으로 생존 분석을 진행합니다. 예제 데이터를 생성하여 Log-Rank Test를 진행해보겠습니다.
import pandas as pd
data = pd.DataFrame({
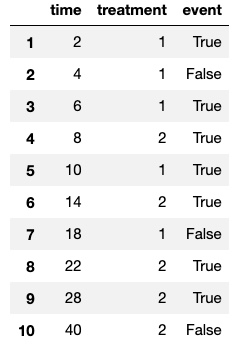
'time' : [2, 4, 6, 8, 10, 14, 18, 22, 28, 40],
'treatment' : [1, 1, 1, 2, 1, 2, 1, 2, 2, 2],
'event' : [True, False, True, True, True, True, False, True, True, False]
}, index = [1, 2, 3, 4, 5, 6, 7, 8, 9, 10])
data총 10개의 샘플 데이터를 생성하였습니다. 데이터는 Time, Treatment, Event 3개의 변수로 구성되며,Treatment 변수는 치료 방법에 따른 생존율을 확인하기 위해 추가하였습니다.
먼저, Kaplan-Meier Curve를 그려 생존율이 어떻게 변화하는지 확인해보도록 하겠습니다. 데이터는 위에서 생성한 예제 데이터를 그대로 사용하겠습니다.
treat1 = data[data['treatment'] == 1]
treat2 = data[data['treatment'] == 2]
from lifelines import KaplanMeierFitter
kmf = KaplanMeierFitter()
kmf.fit(treat1['time'], treat1['event'], label='treat1')
ax_kmf = kmf.plot()
kmf.fit(treat2['time'], treat2['event'], label='treat2')
ax_kmf = kmf.plot(ax = ax_kmf)
ax_kmf.set_xlabel('Time (Months)')
ax_kmf.set_ylabel('Survival Rate')
ax_kmf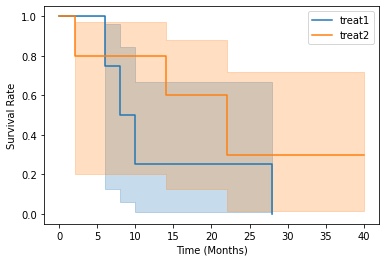
위의 그림을 살펴보면 Treat2이 Treat1에 비해 상대적으로 생존율이 낮은 것 처럼 보입니다. 하지만, 실제로 통계 검정을 하기 전까지 유의한 차이가 있는지 장담할 수 없습니다. 실제로도 유의한 차이가 있는지 확인하기 위해 Log-Rank Test를 해보겠습니다. lifelines 라이브러리가 제공하는 logrank_test 함수를 활용하여 손쉽게 검정을 진행할 수 있습니다.
from lifelines.statistics import logrank_test
logrank_test(treat1["time"], treat2["time"], treat1["event"], treat2["event"]).p_value # 0.3407검정 결과, P-value 는 0.3407로 확인되었습니다. 육안으로는 두 치료 방법이 유의한 차이를 나타내는 것처럼 보였지만, 실제 검정 결과는 달랐습니다. 즉, 두 치료 방법은 유의미한 차이가 없음을 확인할 수 있었습니다.